Une serrure à double combinaison pour neutraliser les gènes sauteurs
Des chercheurs de l’iGReD (UCA/CNRS/Inserm), en collaboration avec l’Université de Cambridge, ont mis au jour un nouveau mécanisme épigénétique permettant de contrôler les « gènes sauteurs », des éléments d’ADN mobiles dont l’activité peut menacer l’intégrité du génome.
Les éléments transposables, parfois surnommés « gènes sauteurs », sont des morceaux d’ADN capables de se déplacer d’un endroit à un autre dans le génome. Présents chez presque tous les êtres vivants, ces éléments ont joué un rôle dans l’évolution, mais ils peuvent aussi devenir dangereux : en bougeant, ils risquent de casser ou perturber le fonctionnement normal de l’ADN. Cela peut provoquer des mutations, altérer certains gènes et favoriser le développement de maladies comme certains cancers.
Le risque est encore plus grand quand ces éléments s’activent dans les cellules germinales (celles qui donnent les ovules ou les spermatozoïdes), car les anomalies peuvent alors être transmises à la descendance.
Pour mieux comprendre comment ces « gènes sauteurs » sont contrôlés, des chercheurs de l’Institut de Génétique, Reproduction et Développement (iGReD, UCA/CNRS/Inserm), en collaboration avec l’Université de Cambridge, ont utilisé la mouche drosophile comme modèle. Ils ont découvert un nouveau mécanisme épigénétique – une forme de régulation qui n'altère pas l’ADN lui-même mais contrôle son activité – permettant de freiner ces éléments mobiles.
Ces résultats ont été publiés dans la revue Nature Structural and Molecular Biology.
Chez la drosophile, les cellules germinales utilisent un système de défense spécifique, conservé de l’éponge de mer jusqu’à l’homme, appelé la voie des piARNs, pour empêcher l’expression des éléments transposables (ET). Cette voie repose sur des ARNs de petite taille et des protéines spécialisées, dont Rhino chez la drosophile, une protéine essentielle à la production des piARNs et à la répression des ET. Sans Rhino les piARNs ne sont pas produits et le contrôle des ETs est perdu. Rhino, une protéine de la famille HP1, est connue pour se lier à certaines régions du génome enrichies en H3K9me3, une marque épigénétique associée à l’hétérochromatine. Toutefois, il restait à comprendre pourquoi elle ne se fixait qu’à une sous fraction des régions portant cette marque.
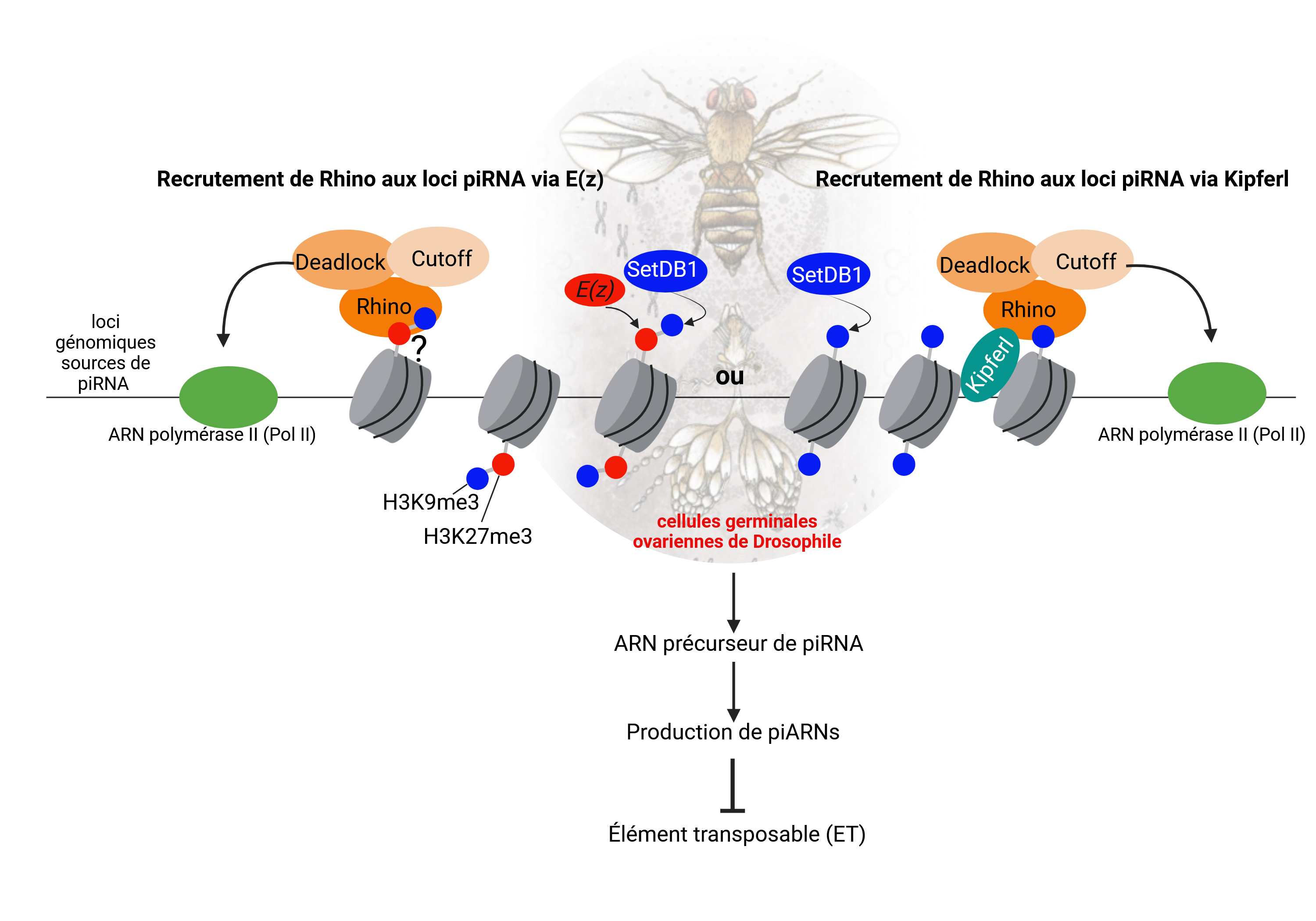
Pour répondre à cette question, les chercheurs ont identifié un nouveau mécanisme impliquant une seconde marque épigénétique, H3K27me3, catalysée par l’enzyme Enhancer of Zeste (Ez). Cette marque, co-localisée avec H3K9me3 sur certains loci spécifiques, forme un "code épigénétique double" qui guide Rhino vers ces régions génomiques précises, les transformant en site de production de piARNs. De manière inattendue, ils ont également montré que ce mécanisme fonctionne indépendamment de Kipferl, une autre protéine qui joue un rôle dans le ciblage de Rhino sur d’autres régions.
Pour tester leurs hypothèses, les chercheurs ont utilisé une combinaison d’approches génétiques, d’imagerie et de séquençage haut débit. Leurs résultats montrent que l’absence d’E(z) et de H3K27me3 perturbe la fixation de Rhino sur certains loci, entraînant une réduction de la production de piRNA et une augmentation aberrante de l’activité des ETs dans les cellules germinales.
Ces travaux soulignent l’importance des mécanismes épigénétiques dans le maintien de la stabilité génomique. En combinant deux marques épigénétiques distinctes, les cellules germinales de la drosophile utilisent une stratégie sophistiquée pour étiqueter spécifiquement les éléments transposables et les réprimer de manière efficace. Ce mécanisme est non seulement crucial pour la drosophile, mais il pourrait également être conservé dans d’autres espèces, soulignant son rôle fondamental.
En conclusion, cette étude met en lumière un exemple remarquable d’interaction entre plusieurs marques épigénétiques pour orchestrer la défense du génome contre les éléments transposables. Ces découvertes ouvrent de nouvelles perspectives pour comprendre comment les modifications de la chromatine régulent la dynamique des génomes au cours du développement et de l’évolution.
© Emilie Brasset
Ce travail a été réalisé en collaboration avec le laboratoire de Greg Hannon au CRUK à Cambridge
Contact chercheuse :
Emilie Brasset
Enseignant Chercheur
emilie.brasset@uca.fr
iGReD (UCA/CNRS/Inserm)
Faculté de Médecine
28 Place Henri Dunant, 63001 Clermont-Ferrand
